ホーム | 統計 Top | JAGSをRから呼び出すパッケージの新定番runjagsについて(190903初版公開)
Martyn Plummer によって開発された JAGS (Just Another Gibbs Sampler) は、BUGS言語を用いて記述された統計モデルに対し、マルコフ連鎖モンテカルロ法によるパラメータ推定を行う簡便なプログラムである。コンピュータにインストールされた JAGS を R から呼び出すツールとして、公式実装である rjags package (Plummer 2019)が伝統的に使われてきた一方、近年着目のパッケージが Matthew Denwood によって開発された runjags (Denwood and Plummer 2019)だ。rjags ではユーザーが手動でインスタンスを立ち上げる必要があった、マルチスレッドによる計算チェーンの並列化が、runjags ではサンプラー関数のオプションとして最初から用意されている。その他、収束診断に基づく計算の自動延長/打ち切りといった、高度かつ省力化された分析機能を有することから、高いユーザーシェアの獲得が予想される。
マルコフ連鎖モンテカルロ法の理論的背景とか、JAGS本体の導入方法とかはすっ飛ばして、いきなりRからJAGSを呼び出す方法の話。ほとんど自分用のあんちょこなので、最初から超実戦的なコードだが堪忍いただきたい。
目次
- とりあえず回帰モデルをJAGSで解いてみる
- 例題:二次関数
- JAGS と runjags を用いた MCMC によるパラメータ推定手順
- run.jags() 関数の出力
- runjags の結果について
- 事後分布の要約表示
- トレースプロット
- JAGSの条件設定
- MCMCの回数の決め方
- マシンスペックと計算時間について
- 初期値リストの作成
- (そのうち書く)収束診断に基づく計算の自動延長/打ち切り
- (そのうち書く)あえてシングルスレッドで計算する
とりあえず回帰モデルをJAGSで解いてみる
例題:二次関数
以下は、 \(\mu=-1-2x+x^2\) という二次関数に従う50個の数値データの組を作り、さらに, \(y = \mathrm{Normal}(\mu=\mu, \sigma=0.5)\) としてランダムにばらつかせている(ただし\(\mu\)、\(\sigma\)はそれぞれ平均、標準偏差)。つまり観測値 y は、二次回帰モデルにおける真の応答値 \(\mu\) に、正規分布する観測誤差を足したものであると考える。
n.occasions <- 50
a.tru <- -1
b.tru <- -2
c.tru <- 1
sd.tru <- 0.5
# coef に入れた任意の長さの数値ベクトルを係数として、多項式の値を出力する。
funcPolynom <- function(x, coef) {
p <- length(coef)
ans <- 0
for (i in 1:p) {
product <- coef[i]*(x^(i-1))
ans <- ans + product
}
return(ans)
}
set.seed(114514) # 皆様のPC環境で同じ結果が得られるように、乱数のシードを指定。
x <- rnorm(n=n.occasions, mean=1, sd=1) # 説明変数の値を作成。
surv15 <- funcPolynom(x, coef=c(a.tru, b.tru, c.tru)) # 応答値 (mu) を作成。
surv15var <- surv15+rnorm(n.occasions, mean=0, sd=sd.tru) # 測定誤差の足された応答値 (y) を作成。
一応データの形を見ておこう。
# 関数の形を描いておく
library(ggplot2)
XLIM <- c(-2, 4)
YLIM <- c(-4, 8)
filename.pdf <- "runjags_Fig01.pdf"
filename.png <- "runjags_Fig01.png"
p <- ggplot(data.frame(x=x), aes(x=x, y=surv15var)) +
stat_function( fun=funcPolynom,
args=list(coef=c(a.tru, b.tru, c.tru)), linetype=1, size=0.75) +
annotate("segment", x=x, xend=x, y=surv15, yend=surv15var, col=rgb(0, 0, 0), size=0.25) +
geom_point(col=rgb(0, 0.5, 0.8), size=0.75) +
geom_abline(intercept=0, slope=0, linetype="solid", size=1/3) +
theme_classic(base_size=9) +
theme(legend.position="none") + # no legend
coord_fixed(ratio=0.5) +
scale_x_continuous(expand=c(0, 0), limit=XLIM, breaks=c(-2, 0, 2, 4)) +
scale_y_continuous(expand=c(0, 0), limit=YLIM, breaks=seq(-4, 8, by=2)) +
annotate( "text", x=1, y=2, label="italic(mu) == -1 -2*italic(x) + italic(x)^2",
parse=TRUE, size=3, hjust=0.5) +
labs( title="", x="x", y="y" )
ggsave(file=filename.pdf, device=cairo_pdf, plot=p, dpi=300, width=8/2.54, height=8/2.54, units="in")
ggsave(file=filename.png, plot=p, dpi=300, width=3, height=3, units="in")
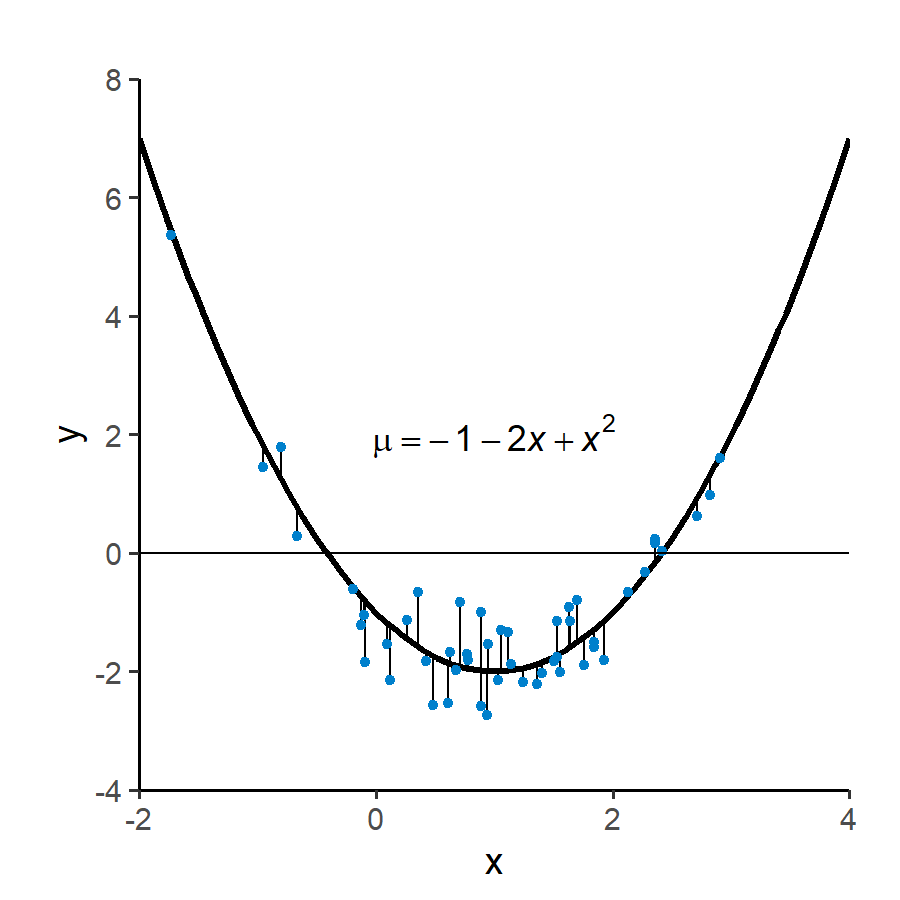
Fig. 1 | 説明変数 x、二次回帰モデルにおける真の応答値 \(\mu\)(曲線)、誤差成分が足された観測値 y(点)の関係。
JAGS と runjags を用いた MCMC によるパラメータ推定手順
説明は後で加えるとして、いきなりMCMCを用いて \(\mu=a+bx+cx^2\) および \( y=N(\mu, \sigma)\)に対応する、パラメータ \(a, b, c, \sigma\) の値を推定してみよう。システムにJAGSがインストール済みで、パッケージとして rjags および runjags がインストールされていれば、上の変数定義とあわせてRコンソールにコピペするだけで手順を再現できるはずだ(環境設定できていなければ下の findjags() で止まる)。
# JAGSに与える観測データ。
jags.data <- list( n=n.occasions,
x=x,
y=surv15var )
# BUGS 言語によるモデル指定。
jagsname <- "model01.jags"
sink(jagsname)
cat("
model {
Tau.nif <- 1.0E-3;
P.gamma <- 1.0E-3;
# 個々のパラメータに対する事前分布
a ~ dnorm(0, Tau.nif);
b ~ dnorm(0, Tau.nif);
c ~ dnorm(0, Tau.nif);
tau ~ dgamma(P.gamma, P.gamma);
sigma <- 1/sqrt(tau);
# 尤度モデル
for (i in 1:n) {
mu[i] <- a + b*x[i] + c*pow(x[i], 2); # x[i]^2 == pow(x[i], 2) in JAGS version 4
y[i] ~ dnorm(mu[i], tau);
}
}
", fill=TRUE)
sink() # sink("ファイル名") から sink() までの内容がカレントディレクトリのファイルに書き込まれる。
# MCMC の条件設定。
monitor <- c("a", "b", "c", "sigma") # 上のモデルで MC のサンプリング対象とする変数。
n.chains <- 3 # チェーンの数は、初期値リスト作成よりも前で定義すること
# stochastic node の初期値リスト。チェーンの数だけ用意する必要があるので、ジェネレータとして作る。
# .RNG.seed と .RNG.name に挟まれた行に、各 stochastic node の初期値を与える乱数を置く。
set.seed(10) # 後日結果を再現したい場合のため、乱数のシードを決めておく。
func_init <- function(i) {
list(
.RNG.seed=i+0.1,
a=rnorm(1, 0, 1),
b=rnorm(1, 0, 1),
c=rnorm(1, 0, 1),
tau=rgamma(1, 1, 1),
.RNG.name="base::Mersenne-Twister")
}
inits <- list(func_init(1)) # 最初の要素を二重リストにしてから、for文内部で append する。
if (n.chains > 1) {
for (i in 2:n.chains) {
set.seed(100+i)
inits <- append(inits, list(func_init(i))) # リストをさらにリストで包んでから連結。
}
}
set.seed(1000);
str(inits); # 一応見ておきましょう
# MCMC の実行。複数コアを使うメソッド "parallel" で runjags する。
name_anl <- "runjags.test101"
library(rjags); library(runjags); findjags(); # パッケージをロード。おまじないで findjags() すべし。
system.time(
assign(name_anl, run.jags( model=jagsname, monitor=monitor, data=jags.data,
inits=inits, n.chains=n.chains,
adapt=1000, burnin=49000, sample=10000, thin=10, # sample*thin 繰り返す
tempdir=FALSE, keep.jags.files=name_anl, method="parallel") )
)
# Graph size 486, chain=3, system time 7.5 sec. # 150000回で7.5秒
get(name_anl) # 事後分布の表示
name_time <- format(Sys.time(), "%Y-%m-%d_%H.%M.%S")
plot(get(name_anl), layout=c(2, 2), file=paste(name_anl, name_time, "pdf", sep="."))
run.jags() 関数の出力
計算中~計算終了時のRコンソールには、以下の内容が出力される。真ん中の 49000 とか 100% とかある場所は、実際にはプログレスバーになっている。
Calling 3 simulations using the parallel method...
Following the progress of chain 1 (the program will wait for all chains to finish before continuing):
Welcome to JAGS 4.2.0 on Sat Aug 31 21:59:47 2019
JAGS is free software and comes with ABSOLUTELY NO WARRANTY
Loading module: basemod: ok
Loading module: bugs: ok
. . Reading data file data.txt
. Compiling model graph
Resolving undeclared variables
Allocating nodes
Graph information:
Observed stochastic nodes: 50
Unobserved stochastic nodes: 4
Total graph size: 468
. Reading parameter file inits1.txt
. Initializing model
. Adaptation skipped: model is not in adaptive mode.
. Updating 49000
-------------------------------------------------| 49000
************************************************** 100%
. . . . . Updating 100000
-------------------------------------------------| 100000
************************************************** 100%
. . . . Updating 0
. Deleting model
.
All chains have finished
Note: the model did not require adaptation
Simulation complete. Reading coda files...
Coda files loaded successfully
Calculating summary statistics...
Calculating the Gelman-Rubin statistic for 4 variables....
Error : The "modeest" package is required to calculate the mode of continuous variables
Finished running the simulation
ユーザ システム 経過
0.63 0.34 7.62
警告メッセージ:
runjags.summaries(fullmcmclist = mcmc, thinnedmcmclist = thinnedmcmc, で:
An unexpected error occured while calculating the mode
runjags の結果について
事後分布の要約表示
計算終了後、パラメータ推定値を表示できる。
> get(name_anl) # 事後分布の表示
JAGS model summary statistics from 30000 samples (thin = 10; chains = 3; adapt+burnin = 50000):
Lower95 Median Upper95 Mean SD Mode MCerr MC%ofSD SSeff AC.100 psrf
a -1.2918 -1.0773 -0.86824 -1.0777 0.10729 -- 0.0006109 0.6 30847 -0.0057313 1
b -2.1074 -1.8752 -1.6437 -1.8764 0.11844 -- 0.00074082 0.6 25561 -0.0068985 1
c 0.85838 0.96282 1.0725 0.9633 0.054349 -- 0.00033898 0.6 25707 -0.0031345 1
sigma 0.41605 0.51317 0.6252 0.51759 0.054536 -- 0.00031486 0.6 30000 0.0012111 1
Total time taken: 5.1 seconds
計算後の出力オブジェクトの名前を打つだけで、複数のチェーンからのサンプリング結果を全自動でまとめた要約統計を返してくれる。本来は mode も計算されるらしいが、なぜか当方の環境で "modeest" package が上手く入らないため省略されている。通常はMCMCの解釈では中央値とクォンタイルを使うため、無くても特に問題はない。
推定されたパラメータの中央値は \(a=-1.0773, b=-1.8752, c=0.96282, \sigma=0.51317\) とある。最初に与えた真のパラメータは \(a=-1, b=-2, c=1, \sigma=0.5\) であったので、b と c が若干ゼロ寄りに偏ってしまうものの、いい線を行っている。
非常にありがたいのは右端の psrf という項目である。これは potential scale reduction factor という MCMC の収束診断指標で、あるパラメータについて複数のチェーンが返すサンプリング結果が共通しているほど、小さな値になる。理想的には 1.0 になるべきで、概ね 1.05 ないし 1.1 以内に収まっていれば、MCMC が収束に達するに十分な計算回数が確保されていたものと解釈される。従来用いられていた rjags パッケージだと、複数チェーンからの結果をまとめて PSRF を計算するための処理を別途コードせねばならず、ここも runjags によって大幅に省力化される点の一つである。
トレースプロット
出力オブジェクトの名前で plot すると、パラメータごとに、複数チェーンの変動を表示したトレースプロット(Fig. 2)が出力される。
plot(get(name_anl), layout=c(2, 2), file=paste(name_anl, name_time, "pdf", sep="."))
このプロットは複数ページになるため、 file="ファイル名.pdf" というフラグを付けて、描画デバイス上での表示ではなく直接ファイルに吐き出させる必要がある。
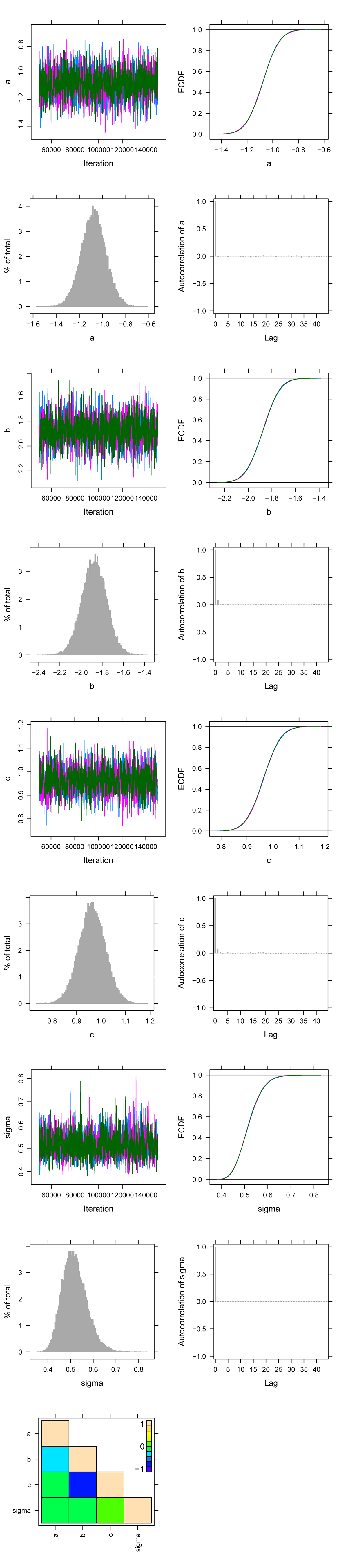
Fig. 2 | 計算後に plot で出力されるトレースプロット。パラメータごとに4枚のプロット+パラメータ間の相関係数、計5ページの pdf を1葉の図にまとめた。
最後のページに、事後分布から推定されたパラメータ間の相関係数(各ステップにおける a, b, c, sigma の値と、別のステップにおける値とを比較している)が示されている。独立変数が本当に独立であれば理論上は相関が 0 付近に収まるはずだが、今回はパラメータ a と b, b と c の間で、強い負の相関を示している。これは \(\mu=a+bx+cx^2\) という関数において、ある次数の係数を変えつつデータにフィッティングさせようとするには、隣接次数の項の係数を変えねばならず、明らかにパラメータ間の関係が独立でないことに由来している。
出力オブジェクトの詳細
なお get(name_anl) という処理は、Rコンソールにおいて runjags.test101 という文字列を "" を付けずに打ち込んだ場合と同じである。通常Rではオブジェクト名を裸で入力すると、オブジェクトの全貌が表示されるのだが、run.jags() の出力は巨大なリストである。幸い、コンソールが埋め尽くされないよう要約値を返してくれるクラス設計になっている。実は各チェーンの生のサンプル値等も保存されているので、興味のある人は names(get(name_anl)) や、str(get(name_anl)) といった、リストの構造を調査する関数を用いて調べてみるとよい。
JAGSの条件設定
MCMC の回数の決め方
JAGSが計算ステップを進める際、最初の adapt 回はマルコフ連鎖ではない(詳しくはJAGSの公式ドキュメントを)ので、そもそもサンプリング対象にならない。次の burnin 回はマルコフ連鎖だが、パラメータの値に初期値の影響が残っている(=収束していない)ため、サンプリング対象としない。次の sample*thin 回が、ひとたび事後分布に達した(であろう)マルコフ連鎖からの、サンプリング対象となるステップ数で、今回であれば各チェーンを10000×10で10万ステップ回しているが、サンプルは10回おきにしか記録しない。つまり、取得されるサンプルサイズは10000×3(チェーン数)×4(モニタリング対象のパラメータ数)である。
この回数調整の基本的な考え方だが、複雑なモデルになるほど収束が遅いので、burnin を多めに取る必要がある。収束したか否かの診断は、最終的には potential scale reduction factor (PSRF) 等の指標を用いるが、実際の調整作業は目視で行うのが早い。まず burnin=0 として初期状態からのパラメータのふらつきを、全てグラフに描いてみる。モデルが適切に組まれていれば、チェーンごとに異なる初期値からスタートした各パラメータの値が、数千ステップ以内に一か所に集まり(Fig. 3A)、以降は同じ場所を中心に小刻みに振動するだけになる(Fig. 2)。これが収束に達したマルコフ連鎖の特徴である。そこで burnin の値を、全てのパラメータの値が定常状態に落ち着くとみられるステップ数よりも多めに設定し、以降をサンプリング対象とする。
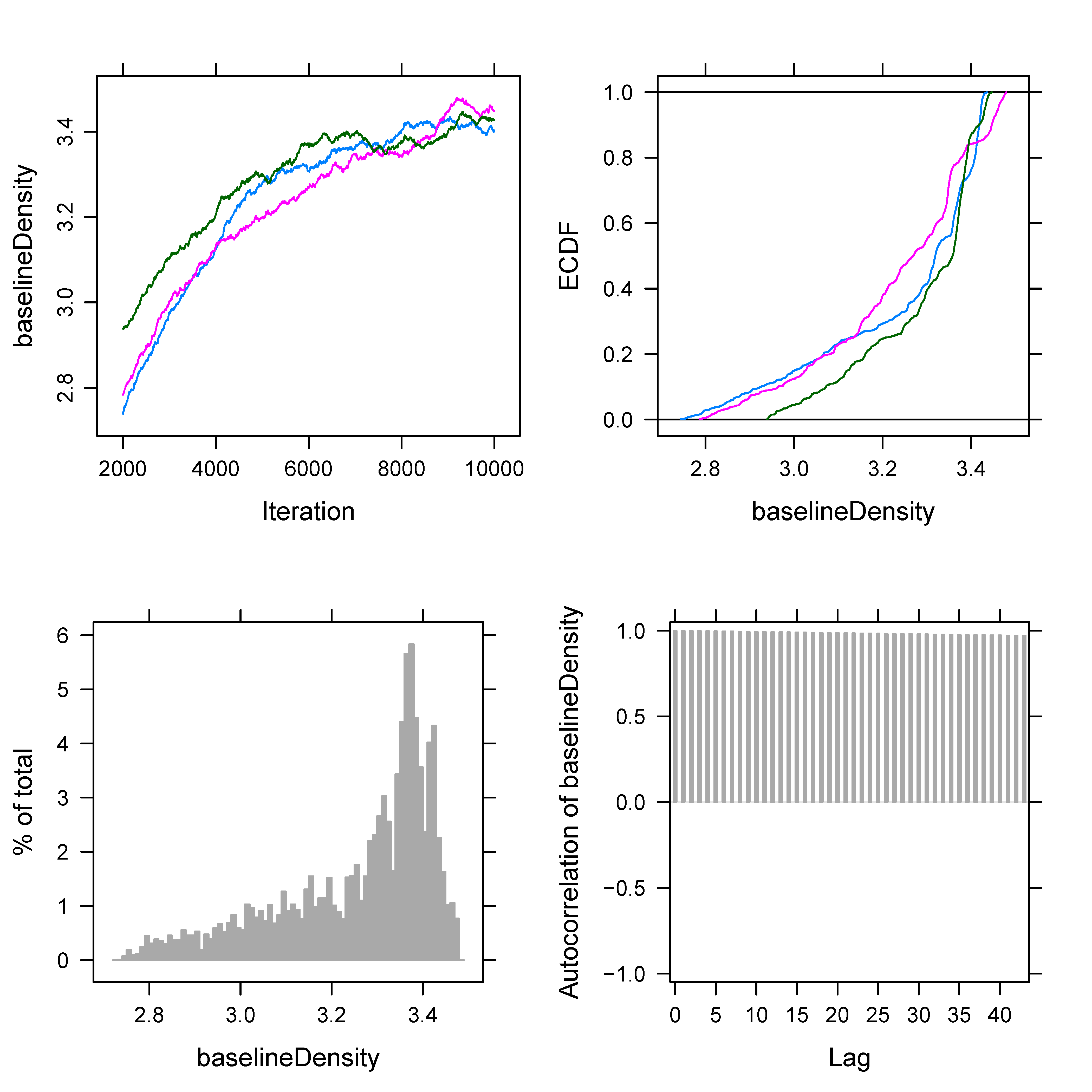
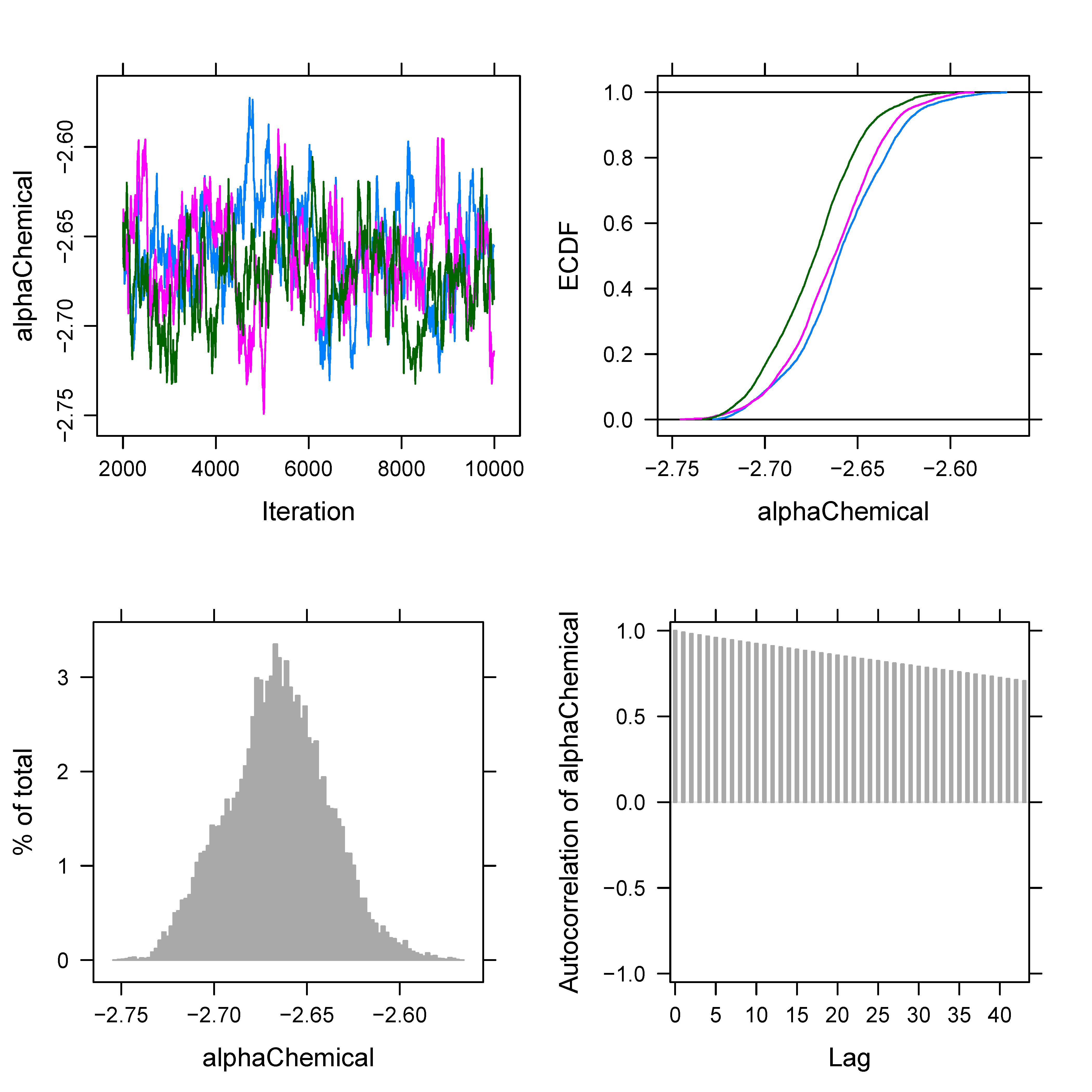
Fig. 3 | (A) 未収束なトレースプロットの例。(B) パラメータの事後分布が一箇所に収束せず、ゆらゆら動き続けるトレースプロットの例。
サンプリング対象とするパラメータ(説明変数)の間に多重共線性(multicollinearity)があると、いったん収束したモデルにおいてもパラメータの値がゆらーりゆらーりと長周期の振動を示すことがある(Fig. 3B)。これは、ある説明変数とその他の説明変数とが、得られるデータの限りにおいて独立でないことを意味する。凄く乱暴なイメージで説明するならば、100円のガムを買うためにタカシ君(説明変数A)とシゲル君(説明変数B)が50円ずつ出すことも可能な一方、タカシ君が10円、シゲル君が90円を出しても買い物には支障がない(片方のパラメータが最適値を逸脱しても、別のパラメータの値を連動してずらすことでモデルの尤度が維持されてしまい、Gibbsサンプラーがなかなか最適値に復帰できない)状態である。これはモデル自体の設計がまずいのだが、あえて結果を出さねばならない場合には、 thin を十分に大きくとることで収束診断値を改善するというテクニックがある。もちろん計算時間は増大してしまう。
マシンスペックと計算時間について
JAGSを用いたMCMCにどの程度の計算時間を要するかは、run.jags() でグラフを初期化した時に表示される Total graph size: 468 という値を見れば概ね類推可能である。今回用いたCPUは Intel Core i7-7700K (基本/最大クロック 4.2/4.5 GHz, 4コア8スレッド)だが、このグラフサイズで15万ステップ計算するのに7.5秒を要している。なお3チェーン分の計算はマルチスレッド化されているので、マシン上の空きスレッドを使い切らない限り(たとえば4コア8スレッドのCPUで、9チェーン以上投入する)、計算時間がチェーン数に比例して増えるには至らない。
それぞれのマルコフ連鎖が定常状態に達している限り、1つのチェーンから n 回サンプリングしても、n 本のチェーンから1回ずつサンプリングしても、理論上は同じ事後分布からのサンプリングであり妥当な手続きである。最近のワークステーション・サーバ向け CPU はコア数の増大が一つのトレンドとなっており、たとえば AMD の Ryzen Threadripper 2990WX は(品薄だけど)本体わずか20万円程度で買えて、実に32コア64スレッドを備える。チェーン 1 本からのサンプリングに1週間かかるとき、チェーン 64 本に増やせば 1 時間半で片付く?
そうは問屋が卸さない。残念だがチェーン数をやたら増やしたところで、それぞれのチェーンが収束するまでに要する時間は減らせない。そもそも MCMC を用いた計算に数日以上の時間が掛かるケースの大半は、モデルの構造が複雑で、Total graph size が数十万、数百万に達する場合であるから、結局は収束前のうろうろが律速段階になってしまうのだ。また複雑なモデルでは上に述べたように、収束後もパラメータの値が長周期振動することがままある。この場合、run.jags() するときの thin、すなわちサンプリング間隔の間引き量を大きくする必要があり、定常状態のチェーンを極端に短くとることが現実的に推奨されない。
では、メニーコアのコンピュータを使う恩恵が全く無いかというと、実は結構あったりする。モデルを開発する段階では、基本的な説明変数の数とか、変数間の交互作用をどのように入れるかとか、ランダム効果をどのように入れるかとか、細かな部分を変えたバリエーションをとにかくたくさん作って実験する必要がある。基本骨格が固定してからも、各パラメータに割り当てる事前分布の形状とか、事前分布の分散の大きさとか、調整を要する事柄が多いため、マシンのコア数は多いに越したことがない。ここで従来の rjags だと、マルチスレッド化が自動で行われないので R のプロセス自体をチェーン数分立ち上げて、なおかつ「同じモデルの複数のチェーン」と「異なるモデルのチェーン」を区別できるよう、ジョブごとの名称を厳密に管理する必要があった。今や runjags があるので、軽い気持ちで CPU の計算能力を極限まで使い倒せる。マシンの中の妖精さんたちは大変である。
初期値リストの作成
上で runjagsの処理コード例を示したが、str(inits) として初期値リストの大まかな形状を見ていた。実際には以下のような形になっている。
> str(inits); # 一応見ておきましょう
List of 3
$ :List of 6
..$ .RNG.seed: num 1.1
..$ a : num 0.0187
..$ b : num -0.184
..$ c : num -1.37
..$ tau : num 0.166
..$ .RNG.name: chr "base::Mersenne-Twister"
$ :List of 6
..$ .RNG.seed: num 2.1
..$ a : num 0.181
..$ b : num 0.785
..$ c : num -1.35
..$ tau : num 2.89
..$ .RNG.name: chr "base::Mersenne-Twister"
$ :List of 6
..$ .RNG.seed: num 3.1
..$ a : num -0.786
..$ b : num 0.0547
..$ c : num -1.17
..$ tau : num 0.389
..$ .RNG.name: chr "base::Mersenne-Twister"
つまり、JAGS における stochastic node =推定したいパラメータごとに初期値を与えたリストを作り、それをチェーンの本数分だけ束ねたリストにしている。この「リストをリストとして束ねる」操作が意外とトリッキーなせいで、先のコード例が複雑(二重リストにしてからアペンド)になっている。なお複数のチェーンはそれぞれ異なる初期値からスタートする必要があるから、現実的な値の範囲でなるべくバラバラな初期値を生成できるように、パラメトリックな擬似乱数をRで生成する関数、すなわち rnorm() や rgamma() を用いている。